Nel processo di conduzione di studi genetici, incontriamo spesso campioni di RNA insufficienti, ad esempio, per studiare minuscoli tumori orali anatomici, anche campioni unicellulari e campioni di mutazioni genetiche specifiche che vengono trascritte a livelli molto bassi nelle cellule umane.Certo, per il test COVID-19, se i tamponi non sono al posto giusto o non sono abbastanza volte durante il campionamento, la dimensione del campione sarà molto bassa, motivo per cui la Commissione di salute e pianificazione familiare è uscita due giorni fa e ha superato il test e se il campionatore di acidi nucleici non ha prelevato sei campioni, puoi segnalarlo.
La sensibilità del reagente è importante perché abbiamo questo o quel problema, quindi cosa possiamo fare per migliorare la sensibilità della RT-PCR?
Prima di discutere le possibili soluzioni, menzioniamo due grandi complicazioni con la situazione che abbiamo appena menzionato.
Prima di tutto, ci preoccupiamo della perdita di RNA quando abbiamo solo poche popolazioni cellulari nel nostro campione.Se vengono utilizzati metodi tradizionali di separazione e pulizia, come il metodo della colonna o il metodo della precipitazione dell'acido nucleico, esiste un'elevata possibilità che i pochi campioni vadano persi.Una soluzione è aggiungere una molecola vettore, come il tRNA, ma anche in questo caso non c'è alcuna garanzia che il nostro esperimento di recupero vada bene.
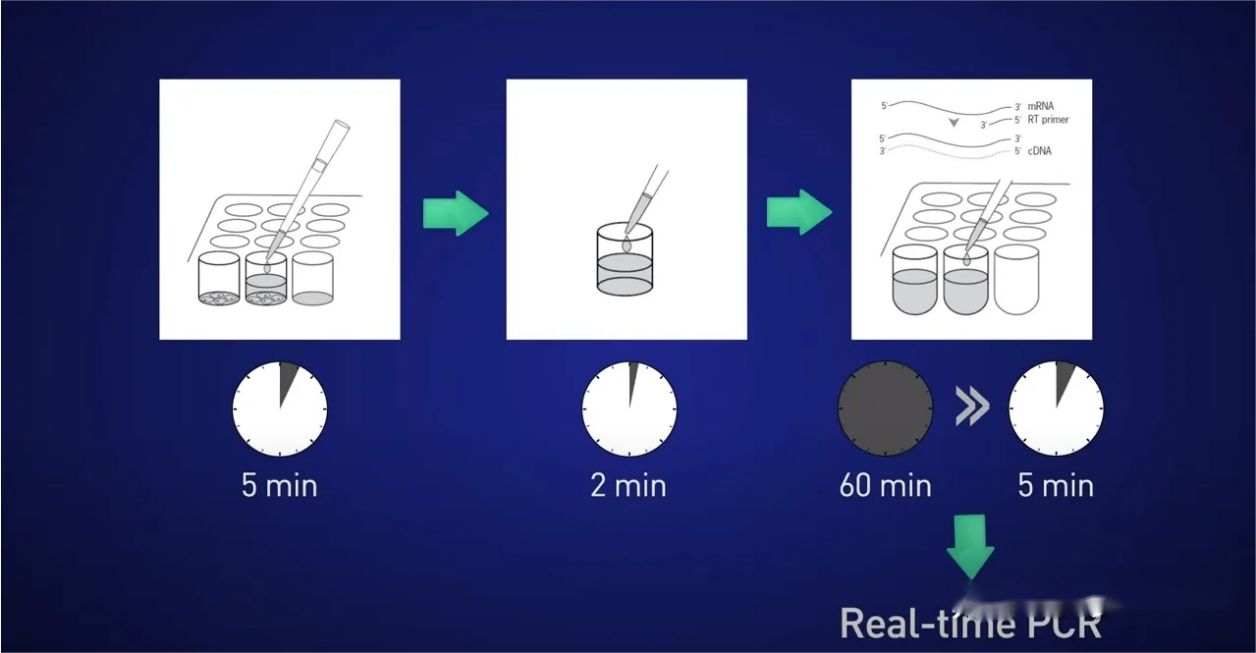
Quindi qual è un modo migliore?Una buona opzione per cellule in coltura o campioni microanatomici consiste nell'utilizzare la lisi diretta.
L'idea è di dividere le cellule per 5 minuti, rilasciare l'RNA nella soluzione, quindi interrompere la reazione per 2 minuti, quindi aggiungere il lisato direttamente alla reazione di trascrizione inversa in modo che nessun RNA venga perso e infine inserire direttamente il cDNA risultante nella reazione in tempo reale.
Ma cosa succede se, a causa di un punto di partenza limitato o di una piccola quantità di espressione genica bersaglio, possiamo riciclare tutto l'RNA e non fornire ancora abbastanza modelli per ottenere un buon segnale in tempo reale?
In questo caso lo step di preamplificazione può essere molto utile.
Quello che segue è uno schema per aumentare la sensibilità dopo la trascrizione inversa.Prima di iniziare, dobbiamo chiedere a valle quali obiettivi siamo interessati, in modo da progettare primer specifici per questi obiettivi per la pre-amplificazione.
Ciò può essere ottenuto creando un primer misto con un massimo di 100 coppie di primer e un ciclo di reazione da 10 a 14 volte.Pertanto, è necessario un Master Mix appositamente progettato per questo requisito per preamplificare il cDNA ottenuto.
La ragione per impostare il numero di cicli tra 10 e 14 è che questo numero limitato di cicli garantisce la casualità tra i vari obiettivi, che è cruciale per i ricercatori che necessitano di informazioni molecolari quantitative.
Dopo la pre-amplificazione, possiamo ottenere una grande quantità di cDNA, in modo che la sensibilità di rilevamento nel back-end sia notevolmente migliorata e possiamo persino diluire il campione ed eseguire più reazioni PCR in tempo reale per eliminare possibili errori casuali.
Tempo di pubblicazione: aprile-11-2023